|
|
 |
 |
|
|
Wolfgang Marks: Die Formatierte DNA
|
|
|
Teil II.
Das programmierte Genom.
Das Genom und sein inhärenter Logos.
Dass die Nukleosomen auf der DNA nicht zufällig angeordnet sind, sondern ihre Positionierung einer Gesetzmässigkeit unterliegt, die zur Transkription, aber auch zur Nicht-Transkription eines Gens führen kann, kann nach dem zuvor Gesagten wohl kaum noch bestritten werden. Wenn es aber eine solche Gesetzmässigkeit, eine solche Ordnung gibt, dann muss es auch biologische Prozesse geben, die diese Ordnung, die Formatierung der DNA mit spezifischen Nukleosomengrössen bewirken.
In diesem Zusammenhang möchte ich den Blick des Lesers auf die sogenannten repetitiven Sequenzen (Interspergiert Repetitive Elemente - IRE) lenken, die in großer Zahl über das ganze Genom verstreut sind. Lange Zeit galten auch sie als „junk-DNA“ – als unnützer Ballast, als Überbleibsel der Evolution59. Auch heute noch sind ihre tatsächlichen Funktionen weitgehendst unbekannt. Nur langsam setzt sich die Erkenntnis durch, dass repetitive Sequenzen für die Regulation der Transkription eine Bedeutung haben könnten. Das NCBI hat deshalb mit der Erfassung und Katalogisierung dieser sowohl in ihrer Struktur als auch in ihrer Länge sehr verschiedenen Sequenzen begonnen.
Grob zusammengefasst lassen sich die Interspergierten Repetitive Elemente (IRE) in diese Gruppen einteilen:
• SINEs = short interspersed nuclear elements
• LINES = long interspersed nuclear elements
• MIR = mammalian wide interspersed repeats
• DNA-Transposons
• Retroelemente
• Pro-/Retroviren
RepeatMasker60 ordnet die repetitiven Elemente wie folgt:
|
SINE
|
LTR Elemente
|
LINE
|
DNA Elemente
|
|
ALU
|
MaLR
|
LINE1
|
MER1_type
|
|
MIR
|
ERVL
|
LINE2
|
MER2_type
|
|
|
ERV_classI
|
L3/CR1
|
|
|
|
ERV_classII
|
|
|
|
An dieser Einteilung werde ich mich orientieren, da sie auch den screenings und Netzwerkanalysen zugrunde liegt, die ich mit diesen Elementen durchgeführt habe.
Nach der klassischen Definition kodieren nur etwa 3 bis 5% des humanen Genoms für Gene. Demnach würden etwa 95% des Erbguts aus Sequenzen bestehen, die keine bekannten Funktionen besitzen. Ein Großteil dieser als funktionslos bezeichneten Sequenzen sind endogene Retroelemente, die mit ca. drei Millionen Kopien mindestens 40 % des humanen Genoms61 ausmachen. Die für Proteine kodierenden Exons der zur Zeit proklamierten 30.00062 Gene des Menschen repräsentieren dagegen gerade einmal 1,5 % des Erbguts. Wenn der Rest, wie vor nicht allzulanger Zeit von durchaus renommierten Wissenschaftlern noch behauptet, tatsächlich „junk“ wäre – also nutzloser Schrott oder Abfall – dann wäre dies ein grandioser Fall von sinnloser Ressourcen-Verschwendung - ein Fall, der in der Natur aber nicht vorkommt.
Aus einer Dissertation von Andreas Herbst, München 2007:
(Zitat) „Nur etwa ein bis zwei Prozent des humanen Genoms bestehen aus kodierenden Sequenzen (Venter, Adams et al., 2001). Dieser kodierende Teil bildet schätzungsweise 20.000 bis 25.000 proteinkodierende Gene. Daneben gibt es viele nicht-proteinkodierende Gene für rRNAs, tRNAs, snoRNAs (small nucleolar RNAs) und miRNAs (micro RNAs). In den letzten Jahren wurde eine große Anzahl neuer Transkripte mittels cDNA-Sequenzierung (complementary DNA) und Micro-Array-Analysen ganzer Chromosomen beschrieben. Schätzungen gehen davon aus, dass ca. 50 % des humanen Genoms transkribiert werden (Semon und Duret, 2004). Micro-Array-Analysen der humanen Chromosomen 21 und 22 haben eine ungefähr zehn mal höhere Transkription ergeben, als im Bezug auf öffentliche Datenbanken erwartet wurde (Kapranov, Cawley et al., 2002).
Erste genomweite Transkriptionsanalysen mit dieser Methode ergaben bei E.coli, dass neben den rund 4.000 bekannten ORFs rund 3.000 weitere im DNA-Gegenstrang (antisense) vorliegen (Selinger, Cheung et al., 2000). Analysen humaner Zellen ergaben außerdem, dass ungefähr zwei mal mehr Nukleotide in polyadenylierten Transkripten repräsentiert sind, als durch Annotierungen öffentlicher Datenbanken zu erwarten war (Kampa, Cheng et al., 2004; Rinn, Euskirchen et al., 2003; Schadt, Edwards et al., 2004). Es wurde gezeigt, dass nur ein sehr geringer Teil der neu beschriebenen Transkripte von neuen proteinkodierenden Genen bzw. von Pseudogenen abstammt. Auch der Versuch, die neuen Transkripte durch alternatives Spleißen oder durch die Verlängerung bekannter Gene zu erklären war nur im einstelligen Prozentbereich möglich (Johnson, Edwards et al., 2005). Ein Teil der neu identifizierten Transkripte zeigt geringe Abundanz und kann nur schwer durch RT-PCR und Northern Hybridisierung nachgewiesen werden (Kampa, Cheng et al., 2004; Rinn, Euskirchen et al., 2003). Außerdem sind nur 7-20 % der neuen Transkripte der humanen Chromosomen 21 und 22 in Maus konserviert, im Gegensatz zu ca. 44 % Konservierung transkribierter Regionen bekannter Gene.“63 (Zitat Ende)
Das Genom: mehr als 80% sind funktionelle DNA.
Was ich 1990 mit dem mir eigenen Verständnis von Natur nur vermuten konnte, kann ich heute mit Argumenten und Daten belegen: das menschliche Genom besteht zum überwiegenden Teil (zu 80% - im weitesten Sinne zu 100%) aus funktionellen Sequenzen. Allein die von mir entdeckten und in dieser Arbeit neu beschriebenen und klassifizierten REMA- und ALU-Gene64 machen zusammen mit den Genen für die mit ihrer Funktion verknüpften UsnRNAs etwa 70% der DNA aus – rechnet man die Gene dazu, die andere RNAs (s-RNA, t-RNA, miRNA u.a.) und Proteine kodieren, kommt man auf ca. 80% funktionelle DNA. Aber auch der Rest ist nicht etwa gänzlich funktionslos, sondern hat wichtige Aufgaben bei der Chromosomenkondensation und der Zellteilung (Telomere, Zentromere, SMARs) und bildet darüberhinaus die „Experimentierküche“, in der ständig an der Verbesserung der bestehenden Strukturen und Abläufe „gearbeitet“ wird.
Besonders überraschend für mich war, dass die Natur – hier vertreten durch den Mikrokosmos Zelle – nicht nur den plus- und den minus-Strang jeweils in der sense-Richtung für die Transkription von RNA und mRNA-Genen verwendet, sondern diese beiden Stränge offensichtlich auch noch in der entgegengesetzten Richtung (reverse/opposit/antisense), also in 5´ <== 3´ bzw. in 3´ ==> 5´ sowohl in extragenischen als auch in intragenischen (intronischen) Territorien zur Produktion von RNAs und Proteinen nutzt, die an der epigenetischen Prägung des Genoms, an der Transkription und an der Regulation der Genexpression entscheidend beteiligt sind.
Beiden von mir entdeckten und in “Die formatierte DNA” erstmals beschriebenen Gensystemen ist eines gemeinsam: sie wären ohne einen inhärenten logos, ohne das zuvor postulierte Nukleosomengrössen-System nicht denkbar. Denn sowohl die REMA-Gene als auch die ALU-Gene bilden ein Netzwerk, in dem die beiden DNA-Stränge und die beiden Transkriptionsrichtungen durch eben diese Nukleosomengrössen zu Adressräumen und damit Informationslayern zusammengefasst sind. Erst die Organisation der Promotoren dieser Gene in Netzwerken mit einer einheitlichen Nukleosomengrösse macht die koordinierte Aktivierung von Promotoren, die koordinierte Transkription und Translation der beteiligten remodeling-Faktoren (REMA-Gene) beziehungsweise Transkriptionsfaktoren (ALU-Gene) möglich.
Im nächsten Abschnitt werde ich versuchen zu zeigen, dass Proteine, die durch Gen-Cluster aus interspergiert repetitiven Elementen (vor allem LINE1-, L3 und MaLR-Elementen) kodiert werden, ein System von remodeling-Faktoren repräsentieren, durch das definierte DNA-Abschnitte (Chromatin-Domänen) während der Mitose mit einer spezifischen Nukleosomengrösse dauerhaft (epigenetisch) „programmiert“ werden.
Interspergiert Repetitive Elemente (IRE): ihre Bedeutung
für die Organisation des Genoms und für die Genexpression.
Repetitive Elemente und hier insbesondere L1-Elemente erfreuen sich seit geraumer Zeit eines gesteigerten Interesses der Forschung: Die Max-Planck-Institute in Berlin zum Beispiel haben in Zusammenarbeit mit der Universität Würzburg unter dem Titel „L1Resources“ ein Internet-Portal geschaffen, das sich ausschließlich mit diesen so lange verachteten („Müll“-DNA) und unterschätzten Sequenzen beschäftigt. Wer sich also ausführlich über Line1-Sequenzen informieren will, findet dort Literaturhinweise und Grundsätzliches über die Funktion von Line1-Elementen als Retrotransposone.
Der folgende Text, den ich ins Deutsche übersetzt habe, stammt von dieser Webseite.
(Zitat Anfang) „Das vollständige Säuger-L1-Element ist ca. 6000 bp lang und besteht aus einer 5´ nicht translatierten Region, in der sich ein interner Promotor befindet, zwei offenen Leserastern (ORF1 und ORF2), die von einer nichtkodierenden Region unterbrochen sind und einem 3´-Bereich mit einer poly-A-Sequenz. Das ORF1 kodiert für ein nicht sequenzspezifisch RNA-bindendes Protein, das ORF2 enthält 3 Domänen, die für die retrotranspositionelle Aktivität verantwortlich sind: eine Endonuklease, eine Reverse Transkriptase und am 3´-Ende eine Zinkfinger-ähnliche Domäne.
Die weitaus meisten L1-Elemente sind in der 5´-Region verstümmelt und /oder enthalten unterschiedliche Insertionen oder Deletionen. Deshalb kommen für eine retrotransposi-tionelle Aktivität in erster Linie auch solche L1-Elemente (FLI-L1) in Frage, bei denen sowohl die zwei Open Reading Frames als auch der im 5´-UTR-Bereich gelegene interne Promotor und die 3´-Region intakt sind. Interessanterweise ist erst vor kurzem vermutet worden, dass L1-Insertionen, die zwar ein unterbrochenes ORF1-Gen, aber ein komplettes ORF2-Gen enthalten (ORF2-L1) imstande sein könnten Alu-Sequenzen zu mobilisieren (Dewannieux et al, Nat Genetics (2003), 35, 41-48).
Eine andere Klasse von L1s, die zur genomischen Funktionalität beiträgt, sind die zwar vollständigen, aber aufgrund vieler Mutationen nicht funktionsfähigen und retrotranspositionell inaktiven L1-Elemente (FLnI-L1). Eine bestimmte Gruppe dieser Elemente könnte die Fähigkeit zur Expression behalten haben und – obwohl mit geringer Frequenz – durch Proteine retrotransponiert werden, die durch intakte retrotranspositionell aktive L1s kodiert werden (Wei et al, Mol Cell Biol (2001), 21, 1429-1439). Ein Teil des regulatorischen Potentials dieser Elemente ist an den im 5´-Bereich lokalisierten Antisense-Promotor gebunden, der, wenn intakt, fähig sein soll, die Expression von Genen zu steuern (Nigumann et al, Genomics (2002), 79, 628-624).“(Zitat Ende)
Die zuletzt in diesem Text zitierten Nigumann et al. schreiben in Genomics 79 (2002):
„Human L1 retrotransposon has two transcription-regulatory regions: an internal or sense promoter driving transcription of the full-length L1, and an antisense promoter (ASP) driving transcription in the opposite direction into adjacent cellular sequences yielding chimeric transcripts. Both promoters are located in the 5'-untranslated region (5'-UTR) of L1. Chimeric transcripts derived from the L1 ASP are highly represented in expressed-sequence-tag (EST) databases. Using a bioinformatics approach, we have characterized 10 chimeric ESTs (cESTs) derived from the EST division of GenBank. These cESTs contained 3' regions similar or identical to known cellular mRNA sequences. They were accurately spliced and preferentially expressed in tumor cell lines. Analysis of the hundreds of cESTs suggests that the L1 ASP-driven transcription is a common phenomenon not only for tumor cells but also for normal ones and may involve transcriptional interference or epigenetic control of different cellular genes.” (Hervorhebung durch den Autor)
Die Übersetzung des letzten (hervorgehobenen) Satzes lautet: „Die Analyse von Hunderten von cESTs lässt vermuten, dass die durch L1 ASP [Antisense-Promotor] angetriebene Transkription ein allgemeines Phänomen ist, das nicht nur in Tumor-Zellen auftritt, sondern auch in normalen Zellen und nicht nur in das Transkriptionsgeschehen, sondern auch in die epigenetische Kontrolle der verschiedensten zellulären Gene involviert sein könnte.“
In der zitierten Arbeit von Niguman et al. über die putative Funktion von Antisense-Promotoren in L1-Elementen ist meines Wissens nichts darüber ausgesagt, auf welche Weise diese die Expression von Genen steuern sollen oder können. Auch wird lediglich von Antisense-Promotoren im 5´-UTR-Bereich gesprochen, Antisense-Promotoren im 3´-UTR-Bereich oder in sonstigen Bereichen der L1-Elemente werden nicht erwähnt.
Da viele der verstümmelten oder verkürzten L1-Elemente im sense-Strang nur noch ein funktionelles ORF oder gar keines mehr aufweisen, ist zumindest für die letzteren die Definition 3´oder 5´UTR a priori obsolet. Hinzu kommt, dass die hier interessierenden L1- (und anderen repetitiven) Sequenzen – wie die erwähnten Studien zeigen - vom plus- und vom minus-Strang auch in entgegengesetzter Richtung transkribiert werden; auch aus diesem Grund sind die Terminologien 5´ bzw. 3´ ohne nähere Erläuterungen nur schwerlich anwendbar oder nachvollziehbar.
Dass Transkripte von L1-Elementen eine Fülle von Spleißstellen aufweisen, ist seit längerer Zeit bekannt. Belancio, Hedges und Deininger65 berichten über extensives Spleissen von L1-RNAs und zwar sowohl von Transkripten des sense- als auch des antisense-Stranges.
Antisense Transkription: nicht die Ausnahme, sondern die Regel.
In neuerer Zeit wird also mehr und mehr deutlich, dass von grossen Teilen des Genoms Antisense-Transkripte von beiden DNA-Strängen generiert werden - antisense-Transkription wird mehr und mehr als „normaler“ zellulärer Prozess verstanden und es werden verstärkt Bemühungen unternommen, die Funktion von antisense-Transkripten bei der Regulation der Genexpression aufzuklären. Lag der Focus der Untersucher bisher auf der sogenannten RNA-Interferenz, z.B dem Ausschalten von Genen mittels RNA-RNA-Basenpaarung, so konzentrieren sich in letzter Zeit die Arbeiten mehr und mehr auf die Rolle von antisense-RNA bei der Modulation der Genexpression selbst:
(Zitat)
“Antisense transcription (transcription from the opposite strand to a protein-coding or sense strand) has been ascribed roles in gene regulation involving degradation of the corresponding sense transcripts (RNA interference), as well as gene silencing at the chromatin level. Global transcriptome analysis provides evidence that a large proportion of the genome can produce transcripts from both strands, and that antisense transcripts commonly link neighboring "genes" in complex loci into chains of linked transcriptional units. Expression profiling reveals frequent concordant regulation of sense/antisense pairs. We present experimental evidence that perturbation of an antisense RNA can alter the expression of sense messenger RNAs, suggesting that antisense transcription contributes to control of transcriptional outputs in mammals.”66
(Übersetzung)
„Antisense-Transkription (Transkription vom entgegengesetzen Strang eines protein-kodierenden oder sense-Strangs67) ist sowohl eine Rolle zugeschrieben worden bei der Genregulation und hier auch bei der Degradation des korrespondierenden sense-Transkripts (RNA-Interferenz) als auch beim silencing von Genen auf der Ebene des Chromatins. Umfassende Transkriptom-Analysen weisen deutlich daraufhin, dass ein grosser Teil des Genoms Transkripte von beiden Strängen generieren kann und dass Antisense-Transkripte häufig benachbarte „Gene“ in komplexen Loci zu Ketten aus verknüpften Transkriptions-einheiten verbinden. Expressionsuntersuchungen zeigen, dass sense/antisense-Paare häufig übereinstimmend (gemeinsam) reguliert werden. Unsere experimentellen Belege zeigen, dass die Unterdrückung einer Antisense-RNA die Expression einer sense-messenger-RNA verändern kann. Es ist deshalb zu vermuten, dass Antisense-Transkription an der Kontrolle der Transkription in Mammaliern beteiligt ist“
Auch Mart Speek vom Center for Gene Technology in Tallin, Estland hat L1-Elemente daraufhin untersucht, ob ihre Antisense-Promotoren (ASP) in die Transkription von Genen involviert sind und vermutet, dass ein erheblicher Teil der L1-Elemente ASPs aufweisen, die mit der Transkription von „normalen“ menschlichen Genen und der epigenetischen Kontrolle von Genen verknüpft sind.68
Das REMA-Gen System -
oder wie aus repetitiven Sequenzen remodeling-Faktoren
und remodeling-Maschinen generiert werden.
Lassen Sie mich noch einmal auf die zuvor erwähnten Untersuchungsergebnisse von Nakamura et al.69 zurückkommen und eine (von mir übersetzte) Passage aus dieser Arbeit zitieren: „Umfassende Transkriptom-Analysen weisen deutlich daraufhin, dass ein grosser Teil des Genoms Transkripte von beiden Strängen generieren kann und dass Antisense-Transkripte häufig benachbarte „Gene“ in komplexen Loci zu Ketten aus verknüpften Transkriptionseinheiten verbinden. Expressionsuntersuchungen zeigen, dass sense/antisense-Paare häufig übereinstimmend (gemeinsam) reguliert werden.„ (Zitat Ende)
Das Genom – also doch nicht nur überwiegend (90%) junk-DNA? Nicht nur nutz- und sinnloser Müll, wie es vor nicht allzulanger Zeit noch allgemeiner Konsens war?
Oder sind die von Nakamura (und anderen Untersuchern) registrierten Transkripte von intra- und extragenischen Sequenzen lediglich funktionslose Artefakte?
Die Suche nach dem Sinn von antisense.
Worin liegen Sinn und Funktion dieser (großen) Zahl von nicht identifizierbaren sense- und antisense-Transkripten? Ich will versuchen, auf diese Fragen eine Antwort zu geben:
Reverse- oder antisense-Transkripte von L1-Elementen (und auch von anderen repetitiven Elementen) wurden bereits in der Vergangenheit immer wieder „verdächtigt“, für Proteine zu kodieren, die mit der Genregulation oder der Vererbung epigenetischer Muster verknüpft sind. Es hat verschiedene Versuche gegeben, durch Erweiterung der Elemente in beide Richtungen zu kodierenden Sequenzen und schließlich zu Proteinen zu gelangen, denen man eine Funktion hätte zuschreiben können. Die Datenbank "Antisense Transcripts with FANTOM2 Clone Set and Their Implications for Gene Regulation"70 ist ein solcher Versuch. Allerdings hat man in dieser Datenbank offensichtlich nur Sequenzen berücksichtigt, die für mehr als 100 Aminosäuren kodieren - ein Ansatz, der meines Erachtens nicht zum Erfolg führen kann.
Ich bin einen anderen Weg gegangen: angeregt durch verschiedene Arbeiten über das Protein NUMA (syn. NUMA1) habe ich mit Hilfe von IMPACD® nach potentiellen DNA-Bindestellen für eine bestimmte Domäne dieses „Nuclear-Mitotic-Apparatus- Proteins“ gesucht, einem Protein, dem sowohl eine wichtige Rolle bei verschiedenen Vorgängen im Rahmen der Mitose als auch bei der Organisation des Chromatins zugeschrieben wird. Das in NCBI unter der GeneID 4926 gelistete Protein soll aus 2115 Aminosäuren bestehen und verbunden mit dynein und dynactin eine wichtige Rolle bei der Ausbildung der Mikrotubuli und bei ihrer Anbindung an die Spindelpole während der Zellteilung spielen (Hsin-Ling Hsu et al.)71. NUMA soll in Interphase-Zellen aber auch ein wichtiger Bestandteil der nuklearen Matrix und an der Reorganisation des Chromatins nach der Mitose beteiligt sein.
Die im Zusammenhang mit der Organisation der REMA-Gene und der Steuerung ihrer Transkription zweifellos interessantere Arbeit aber ist die von Abad, Patricia et al.72, die sich mit dem Einfluss dieses Proteins auf die Organisation des Chromatins auseinandersetzt.
Aus dem abstract (Zitat):
„A potential role for NuMA in nuclear organization or gene regulation is suggested by the observations that its pattern of nuclear distribution depends upon cell phenotype and that it interacts and/or colocalizes with transcription factors. To date, the precise contribution of NuMA to nuclear function remains unclear. Previously, we observed that antibody-induced alteration of NuMA distribution in growth-arrested and differentiated mammary epithelial structures (acini) in three-dimensional culture triggers the loss of acinar differentiation. Here, we show that in mammary epithelial cells, NuMA is present in both the nuclear matrix and chromatin compartments. Expression of a portion of the C terminus of NuMA that shares sequence similarity with the chromatin regulator HPC2 is sufficient to inhibit acinar differentiation and results in the redistribution of NuMA, chromatin markers acetyl-H4 and H4K20m, and regions of deoxyribonuclease I-sensitive chromatin compared with control cells. Short-term alteration of NuMA distribution with anti-NuMA C-terminus antibodies in live acinar cells indicates that changes in NuMA and chromatin organization precede loss of acinar differentiation. These findings suggest that NuMA has a role in mammary epithelial differentiation by influencing the organization of chromatin.“ (Zitat Ende)
Abad et al. konnten also nachweisen, dass NUMA in epithelialen Zellen der weiblichen Brust sowohl in der nukleären Matrix als auch in den sogenannten Chromosomen-Kompartimenten vorhanden ist. Die Expression eines Teil des C-terminus des NUMA-Proteins reichte aus, um die Differenzierung der azinären Zellen zu verhindern und führte in der Folge zu einer Redistribution sowohl des NUMA-Proteins als auch von prägnanten Histon-Markern und von DNAse-I –sensitiven Stellen. Abad et al. schließen deshalb aus ihren Untersuchungen, dass NUMA –Proteine eine wichtige Rolle bei den Differenzierungsvorgängen in den epethelialen Zellen der weiblichen Brustdrüse spielen.
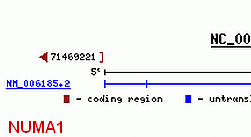 Das abgebildete polycistronische Gen NUMA kodiert nach meinen Analysen ingesamt 27 verschiedene Proteine. Von diesen 27 gehören vermutlich 7 zu einer aus insgesamt 19 Mitgliedern bestehenden Proteinfamilie, deren Mitglieder ausser durch das NUMA-Gen noch durch drei weitere polycistronische Gene kodiert werden. Jedes dieser 19 Proteine ist mit einem bestimmten transkriptionsaktiven Formatierungshormon korreliert. Von den hier gezeigten Transkripten sind die Splicevarianten BC008345.1 und BC013023.1 nach meinen Berechnungen Mitglieder dieser Proteinfamilie und lassen sich deshalb systematisch bestimmten Formatierungshormonen zuordnen. BC008345.1 entspricht in meinem System dem Protein NUMA248 und BC013023.2 (NUMA1) dem Protein NUMA252. Das bedeutet, dass diese beiden Proteine mit diesen Nukleosomengrössen bzw. mit den diesen Grössen zugeordneten Formatierungshormonen GH-RH bzw. Methionin-Enkephalin verknüpft sind. Das abgebildete polycistronische Gen NUMA kodiert nach meinen Analysen ingesamt 27 verschiedene Proteine. Von diesen 27 gehören vermutlich 7 zu einer aus insgesamt 19 Mitgliedern bestehenden Proteinfamilie, deren Mitglieder ausser durch das NUMA-Gen noch durch drei weitere polycistronische Gene kodiert werden. Jedes dieser 19 Proteine ist mit einem bestimmten transkriptionsaktiven Formatierungshormon korreliert. Von den hier gezeigten Transkripten sind die Splicevarianten BC008345.1 und BC013023.1 nach meinen Berechnungen Mitglieder dieser Proteinfamilie und lassen sich deshalb systematisch bestimmten Formatierungshormonen zuordnen. BC008345.1 entspricht in meinem System dem Protein NUMA248 und BC013023.2 (NUMA1) dem Protein NUMA252. Das bedeutet, dass diese beiden Proteine mit diesen Nukleosomengrössen bzw. mit den diesen Grössen zugeordneten Formatierungshormonen GH-RH bzw. Methionin-Enkephalin verknüpft sind.
Meine eigenen Analysen haben ergeben, dass das NUMA-Gen eine Vielzahl von Proteinen kodiert, von denen einige – wahrscheinlich sieben - zu einer Proteinfamilie gehören, die insgesamt 19 Proteine umfasst – und damit wiederum gerade ebenso viele Proteine, wie es in meinem System transkriptionsaktive Formatierungshormone und mit ihnen verknüpfte Nukleosomengrössen gibt. Die Proteine dieser Familie binden an etwa 7 bis 15 Basenpaare umfassende Sequenzen innerhalb von SMARs der Klasse II73 und markieren dadurch unter anderem Anfang und Ende von Transkriptionsabschnitten, die ich aus Gründen, die ich nun näher erläutern werde, REMA-Gene genannt habe.74
|
|
|
59Ich habe schon 1991 in meiner verschiedentlich erwähnten Arbeit die Hybris moniert, die darin liegt, dass man etwas als „junk“ bezeichnet, dessen Funktion man nicht erkennt. Diese Art Hybris ist im übrigen auch heute noch weit verbreitet: alles, was über den jeweils vorhandenen naturwissenschaftlichen Horizont hinausgeht, wird nur allzugern als unwissenschaftlich disqualifiziert.
60Smit, AFA, Hubley, R & Green, P. RepeatMasker Open-3.0. 1996-2004 <http://www.repeatmasker.org>.
61Ich würde meinen, dass es eher 60% sind. Ein erheblicher Teil dieser Elemente wurde noch gar nicht entdeckt.
62Diese Zahl schwankt ständig. Es gibt auch Schätzungen, die bis 80000 gehen.
63“Molekularbiologische Analysen zur Regulation des T-Zell-Rezeptor-b-Lokus“ von Andreas Herbst, Dissertation zur Erlangung des Doktorgrades der Fakultät für Chemie und Pharmazie der Ludwig-Maximilians-Universität München, 2007
64Die ALU-Gene werden in Teil II diese Arbeit besprochen.
65Victoria P. Belancio, Dale J. Hedges, Prescott Deininger: LINE-1 RNA-splicing and influences on mammalian gene expression. Nucleic Acids Research, 2006, Vol. 34, No. 5
66“Antisense Transcription in the Mammalian Transcriptome”, Nakamura et al. Science, 2 September 2005: Vol. 309. no. 5740, pp. 1564 - 1566
67Unterstellt, ich habe den englischen Text richtig verstanden und übersetzt, ist diese Definition von antisense-Transkription wohl falsch oder zumindest ungenau und mißverständlich.
68Antisense Promoter of Human L1 Retrotransposon Drives Transcription of Adjacent Cellular Genes, Molecular and Cellular Biology, March 2001, p. 1973-1985, Vol. 21, No. 6
69“Antisense Transcription in the Mammalian Transcriptome”, Nakamura et al. Science, 2 September 2005: Vol. 309. no. 5740, pp. 1564 - 1566
70http://genome.gsc.riken.go.jp/m/antisense/
71Dynamic changes of NuMA during the cell cycle and possible appearance of a truncated form of NuMA during apoptosis, Hsin-Ling Hsu and Ning-Hsing Yeh Journal of Cell Science, Vol 109, Issue 2 277-288
72NuMA influences higher order chromatin organization in human mammary epithelium.
Abad, Patricia C, Lewis, Jason, Mian, I Saira, Knowles, David W, Sturgis, Jennifer, Badve, Sunil, Xie, Jun, Lelièvre, Sophie A - Journal: Mol Biol Cell, 18 (2): 348-61 2007
73SMAR – Abkürzung für Scaffold/Matrix Attachment (oder Associated) Region. Zur Einteilung der SMARSs in Klassen oder Typen gebe ich weiter unten noch Erläuterungen.
74Ich habe viel Zeit mit den Versuchen verbracht, die tatsächliche Größe eines REMA-Gens zu bestimmen. Der Weg, über cDNA-, EST- oder Protein-Datenbanken potentielle Genprodukte von REMA-Genen zu entdecken, schien mir aussichtlos, da die von mir postulierten remodeling-Maschinen aus dynamischen Proteinkomplexen bestehen sollten, deren einzelne Faktoren durch ORFs verschiedener REMA-Gene kodiert werden. Es konnten deshalb auch nur wenige oder keine ORFs existieren, die Proteine von 120, 130 oder mehr Aminosäuren kodieren, sondern es sollte stattdessen mehrere kleine ORFs geben, die für entsprechend kurze Polypeptidketten kodieren und deshalb erst nach dem Spleißen funktionelle mRNAs ergeben. Solche Transkripte von sehr kurzen ORFs sind aber mit herkömmlichen Methoden kaum zu identifizieren – vielleicht mit ein Grund, warum man sie bisher nicht entdeckt hat (oder sie zwar entdeckt, aber ihre Funktion nicht erkannt hat). Die Identifizierung von REMA-Genen ist nicht zuletzt deshalb überaus zeitraubend und mühsam, weil die zur Verfügung stehenden Programme (RepeatMasker zum Beispiel) eine vorgegebene Sequenz (zum Beispiel eine Chromatin-Domäne) am 5’-Ende beginnend neu durchnumerieren, anstatt von den ja vorhandenen chromosomalen Koordinaten ausgehend zu numerieren. Auch ENSEMBLE zeigt in der ContigView zwar die bis dato bekannten Repeats, nicht aber zu welchem DNA-Strang sie gehören. Dies und die zuvor erwähnte Art des Numerierens erschwert die Einordnung der von RepeatMasker identifizierten Repeats in einen chromosomalen Kontext ungemein.
|
|
|
|